微生物の酵素が生産するバイオ燃料 ~「バイオガソリン」の生産を目指して~
[紹介論文] 1) M. A. Rude et al., Appl. Environ. Microbiol. 2011, 77, 1718-1727. 2) J. Belcher et al., J. Biol. Chem. 2014, 289, 6535-6550. 3) J. L. Grant et al., Proc. Natl. Acad. Sci. U. S. A. 2016, 113, 10049-10054. 4) Y. Liu et al., Biotechnol. Biofuels 2014, 7, 28. 5) A. Dennig et al., Angew. Chem. Int. Ed. 2015, 54, 8819-8822. 6) C. Lu et al., ACS Catalysis 2018, 8, 5794–5798. 7) H. Onoda et al., Catal. Sci. Technol. 2018, 8, 434-442.
この記事の見出し
はじめに
石油や石炭、天然ガスといった化石燃料は、安価な第一次エネルギー源として、経済の発展を著しく加速しました。
昔から、化石燃料は枯渇すると取り沙汰されてていますが、科学技術の発展により、化石燃料の埋蔵地が新しく発見されたり、シェールガスやメタンハイドレードといった新しい燃料が発見されているので、直ちに、エネルギーが枯渇するといったことは無いと、筆者は考えています。
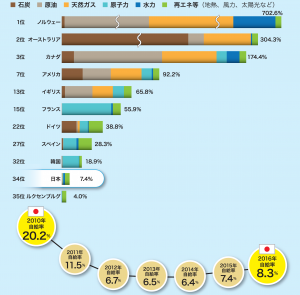
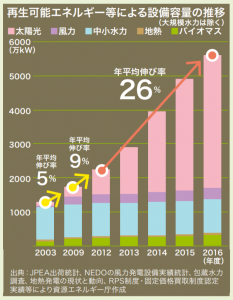
しかしながら、日本のエネルギー自給率は8.3%(2016年現在)と低い割合であるため、日本経済は化石燃料の価格によって大きく左右されてしまうという問題を抱えています。近年、風力や太陽光を含む再生可能エネルギーが普及し、エネルギー自給率が最低であった2014年(6.4%)から約2%改善しています。
私は、再生可能なエネルギーの一つであるバイオ燃料(バイオマス)の開発にも取り組んでいます。ここでは、植物油から生産された脂肪酸エステルとは異なる、酸素原子を含まない「バイオディーゼル」について、さらに、酵素が生産する新しいバイオ燃料「バイオガソリン」に関して紹介致します。
バイオディーゼル
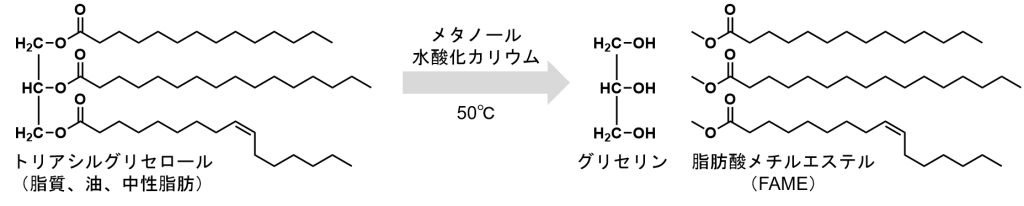
植物油(トリアシルグリセロール、脂肪酸トリグリセリド)からエステル交換反応で生産されるバイオディーゼルは、1892年にディーゼルエンジンを発明したルドルフ・ディーゼルによって、ピーナッツ油由来のバイオディーゼルで動くディーゼルエンジンを発表した頃(1900年)から存在する、最古のバイオ燃料です。
脂肪酸メチルエステル(FAME)を利用したバイオディーゼルは、世界各国で利用されており、最も認知されているバイオ燃料です。日本では、100%FAMEのバイオディーゼル、もしくは、揮発油等の品質の確保等に関する法律の基準を満たした最大5%FAME含有の軽油がディーゼル車の燃料として認可されています。
脂肪酸メチルエステルの性質上、エステルが加水分解される事で酸性度が上昇したり、軽油由来のステロールと共存下で結晶化される事ででフィルターが詰まったり、揮発特性の違いによりエンジンオイルが燃料希釈されたり、品質が軽油とは大きく異なる為、FAMEはディーゼル車に負荷がかかるという問題がありました。
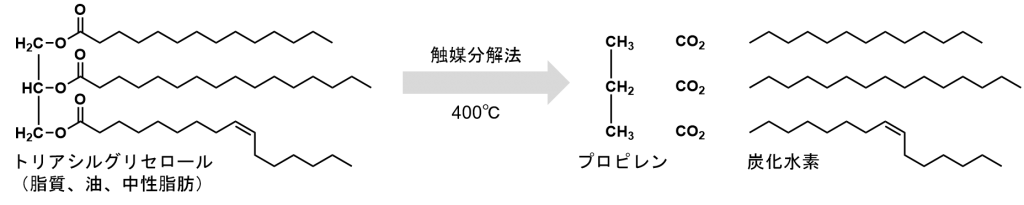
近年では、植物油を水素化したバイオディーゼル(HBD、HVO、BHD®)や、接触分解によってエステルを脱炭酸反応で取り除いたバイオディーゼルHiBDのように、炭化水素を主原料する次世代バイオディーゼルの開発が取り組まれています。この手法で生産されたバイオディーゼルは、化石燃料由来の軽油と構成要素が似ており、軽油の品質を低下させないので、軽油と混ぜた際の制限がありません。
2007年、トヨタ自動車では、新日本石油や日野自動車と合同で東京都でBHDで駆動するハイブリッド(HV)バスの運行を行い、2010年には、化石燃料の代替燃料として期待されているFTD燃料とBHDの混合燃料で駆動するハイブリッド(HV)バスの運行にも取り組んでいたようです。
バイオガソリン
「バイオガソリン」とGoogle検索すると、バイオETBE配合ガソリンという、ガソリンに対して、数%のターシャリーブチルエチルエーテル(ETBE)を混合させた混合燃料が、検索の上位に表示されます。
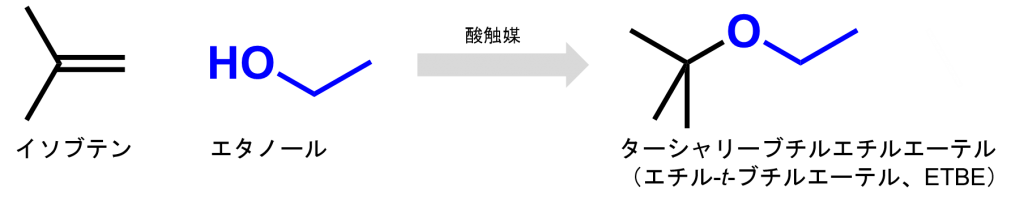
バイオETBEはバイオエタノールと石油成分のイソブテンから合成されており、再生可能なバイオ由来の成分の構成率は44%程度です。近年、フランスのIBN-One社が発酵法でバイオイソブテン生産プラントの建設に取り組んでおり、バイオETBEが100%生物由来の資源で生産される時代が来るかもしれません。
しかしながらETBEは、全分子量に対して16質量%の酸素原子を含むので、NOxを増加させる要因となるガソリン中の酸素分(最大 1.3質量%、E10対応ガソリン車の場合は最大3.7質量%)を高くしてしまうという問題があります。揮発油等の品質の確保等に関する法律に従うと、ETBEは最大8.3%までしか添加する事ができません。イソブテンが化石燃料由来の成分をあること考慮すると、バイオETBE配合ガソリンの再生可能な要素は、現状、4%にも届きません。
バイオETBE含有ガソリンは、ガソリンの代替燃料ではなく、バイオ由来の成分で最大4%嵩増しされたガソリンとなります。
このトピックレビューの目的
このトピックレビューでは、バイオETBEのようなガソリンの添加物を作る手法ではなく、第二世代のバイオディーゼルのように、軽油やガソリンの主成分である「炭化水素」を、微生物に多く含まれる脂肪酸から、微生物由来の酵素で生産する取り組みを紹介します。

CYP152酵素が触媒する脂肪酸から炭化水素の生産反応
シトクロムP450(CYP)は、1958年に450 nmの光(紫~青)を吸収するヘムタンパク質として、大村 恒雄先生によって1962年に名付けられたヘム–チオレート酵素です[1]。翌年、Estabrookらによって、「シトクロムP450は酸化反応を触媒する機能を持つ」と報告されました[2]。CYPの発見に日本人が大きく貢献した経緯から、日本ではシトクロムP450の研究が活発に行われています。
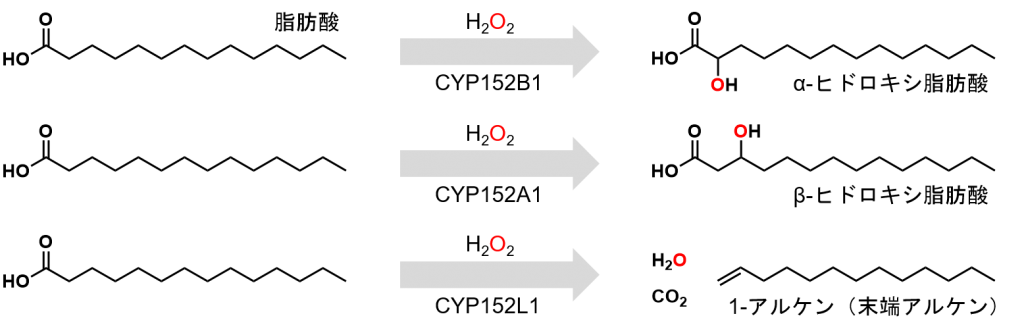
1994年、松永 勇先生に発見された152ファミリーに属するCYP152酵素(CYP152B1[3])は、生体内の過酸化水素を利用して脂肪酸のα-位水酸化反応を行う酵素です[4]。CYP152B1を保有するスフィンゴモナス・パウシモビリス(Sphingomonas paucimobilis)の細胞膜は、人の細胞膜にも含まれるスフィンゴ脂質を含み[5]、このスフィンゴ脂質を構成する脂肪酸のα-位はCYP152B1により水酸化されていると考えられております。
1999年には、枯草菌(Bacillus subtilis)由来のCYP152A1が、脂肪酸のα-位だけでなく、β-位を水酸化出来ると、松永先生らによって報告され[6]、2003年には、城 宜嗣先生らによって、初めてCYP152酵素の構造が明らかにされました[7]。私が所属している渡辺 研究室では、城 研究室と協力し、CYP152B1の結晶構造[8]やCYP152N1の結晶構造[9]の解析に成功しております。
2011年、炭化水素合成菌の Jeotgalicoccus sp. ATCC 8456から発見されたCYP152L1(OleTJE)は、脂肪酸の水酸化反応だけでなく、脂肪酸のβ-酸化的脱炭酸反応を触媒し、炭化水素の一種である「末端アルケン」を「脂肪酸」から生産できると報告されました[10]。この報告の後、CYP152ファミリー酵素はバイオガソリンの生産酵素として活発に研究されるようになりました。
以降、CYP152L1によるアルケン生成のブレイクスルーとなった報告を数件紹介致します。
末端アルケン生成菌(Jeotgalicoccus)の発見とその経緯
CYP152L1を発見したRudeらは、大腸菌にアシル-ACPレダクターゼとアルデヒドデカルボニラーゼを発現させることで、1Lの大腸菌培養液当たり0.1 gのアルカンを生成させることに成功し、2010年にNature誌で報告しています[11]。
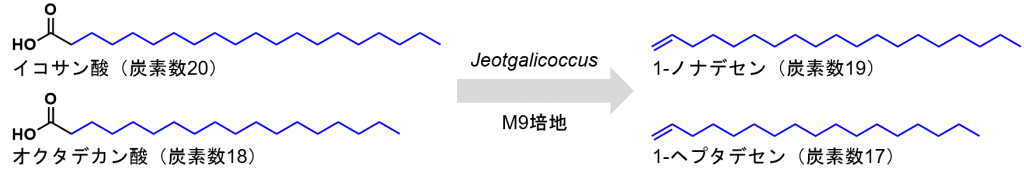
1971年には、炭素数16から22程度のアルケンを生成する菌としてMicrococcus candicans ATCC 8456が報告されていました[12]。この菌に生産される「アルケンの詳細な構造」が判明していなかったため、RudeらはMicrococcus candicans ATCC 8456のアルケンを分析しました。驚くべきことに、検出されたアルケンは全て末端アルケンで、その約80%が炭素数20の末端アルケンでした。
Micrococcus属のDNAのGC含量は65〜75%と知られていますが、Micrococcus candicans ATCC 8456のGC含量は37%であったため、Rudeらは、末端アルケンを生成する菌がMicrococcus属の菌ではないと判断しました。16S rRNA系統解析をおこなうと、100%信頼限界でJeotgalicoccus属の菌として判断され、Jeotgalicoccus marinus JSM 076033 [13] や Jeotgalicoccus sp. strain NY-2 [14]と99%の配列相同性を示しました。この菌が保有する脂肪酸がJeotgalicoccus 属の傾向とよく似ていることからも[15]、この末端アルケンを生成する菌を、Jeotgalicoccus sp. ATCC 8456と再分類すべきと結論づけました。
実際に、Jeotgalicoccus属の菌(Jeotgalicoccus halotolerans DSMZ 17274, Jeotgalicoccus psychrophilus DSMZ 19085, and Jeotgalicoccus pinnipedialis DSMZ 17030)が保有する70%以上のアルケンは、炭素数20の末端アルケンであると確認されました。
炭素数20の長鎖脂肪酸(イコサン酸)を含む培地で生育したJeotgalicoccus sp. ATCC 8456は、炭素数19の末端アルケン(1-ノナデセン)を主なアルケンとして保有し、炭素数18の長鎖脂肪酸(オクタデカン酸)を含む培地で生育した菌は、炭素数17の末端アルケン(1-ヘプタデセン)を生産しました。この結果から、Jeotgalicoccus sp. ATCC 8456は脂肪酸の脱炭酸反応を行い、原料の脂肪酸よりも炭素数が1少ない末端アルケンを生成している事が分かりました。
CYP152L1の発見
Jeotgalicoccus sp. ATCC 8456の細胞抽出液に対して脂肪酸を添加した場合でも、末端アルケンが得られたという結果から、大腸菌抽出液の中に脂肪酸の脱炭酸酵素が溶出している事が分かりました。この細胞抽出液を様々な濃度の硫酸アンモニウムで塩析(硫安沈殿)させると、50%から65%硫酸アンモニウムで塩析された画分に脂肪酸の脱炭酸酵素が含まれている事が分かりました。さらに、強陽イオンカラム(SP)と強陰イオン交換カラム(Q)を用いて精製を行うと、約50kDaと約15kDaのタンパク質を含む、脂肪酸の脱炭酸活性を示す画分が得られました。
Jeotgalicoccus sp. ATCC 8456のオープンリーディングフレーム(ORF)解析を行うと、2.1 Mbpの塩基配列中に1,584個の発現可能なタンパク質が確認されました。880番目のORFが翻訳された場合に発現する48,367Daのタンパク質が、脂肪酸の脱炭酸反応を触媒する酵素であると推定されました。
ORF880の遺伝子を含むプラスミドを用いて形質転換された大腸菌は、炭素数15の末端アルケン(1-ペンタデセン)と、炭素数17のジエン(1-10-ヘプタデカジエン)を生産しました。通常の培養で、この大腸菌は炭素数17の末端アルケンを生成しませんでしたが、炭素数18の脂肪酸(オクタデカン酸)を添加して、この大腸菌を培養すると、炭素数17の末端アルケンを生成することが分かりました。この結果から、ORF880に由来するタンパク質が脂肪酸の脱炭酸反応を触媒している事が決定しました。
ORF880がコードするタンパク質は、CYP152ファミリー酵素と59%のアミノ酸相同性を持つ事が示されました。そこで、6種類のCYP152ファミリー酵素を大腸菌で発現し、精製酵素の触媒活性を調べると、4種類のCYP152酵素は脂肪酸の脱炭酸反応を触媒できることが判明しました。この結果から、脂肪酸の脱炭酸反応はCYP152ファミリーが広範に保有し得る機能であると考えられています。
この論文の著者らは、オレフィン合成酵素群OleABCDにちなんで、この脂肪酸脱炭酸酵素に「OleTJE」という慣用名をつけていますが、次の項で紹介するMunroらは、NelsonらのCYP系統名に従い、この酵素を「CYP152L1」として登録しています。以後、この脂肪酸脱炭酸酵素はCYP152L1という表記で統一させていただきます。
【蛇足】
星の名前と同じように、タンパク質の慣用名は第一報告者によって命名される事が多いです。この報告以前のCYP152ファミリー酵素は、「P450+菌体名の頭文字(+脂肪酸の酸化位置)」という傾向の元命名されていました(例:Sphingomonas paucimobilis由来のCYP152B1(P450SPα)、Bacillus subtilis由来のCYP152A1(P450BSβ)、Clostridium acetobutylicum由来のCYP152A2(P450Cla)[16])。私が報告したExiguobacterium sp. AT1b由来のCYP152N1は、既報に倣ってP450Exαと命名しました[9]。OleTJE命名以降、CYP152P1のCYP-MP[17]や、CYP152L2のCYP-Sm46[18]といった慣用名のCYp152ファミリー酵素も報告され、ファミリー内で慣用名の統一感が無くなってしまいました。私の論文中ではアミノ酸相同性を反映した系統名を用いて表記しています。
結晶構造の解析
酵素の触媒機構を原子レベルで解明する上で、酵素の構造を明らかにすることは重要な課題です。
すでに、CYP152ファミリー酵素であるCYP152A1やCYP152B1の結晶構造が報告されていましたので、「脂肪酸のカルボキシル基が、ヘムに配位結合した過酸化水素の一般酸塩基触媒として機能することで、ヘムの酸化活性種が生成し、ヘムの酸化活性種により脂肪酸が水酸化される」という触媒機構が知られていました。
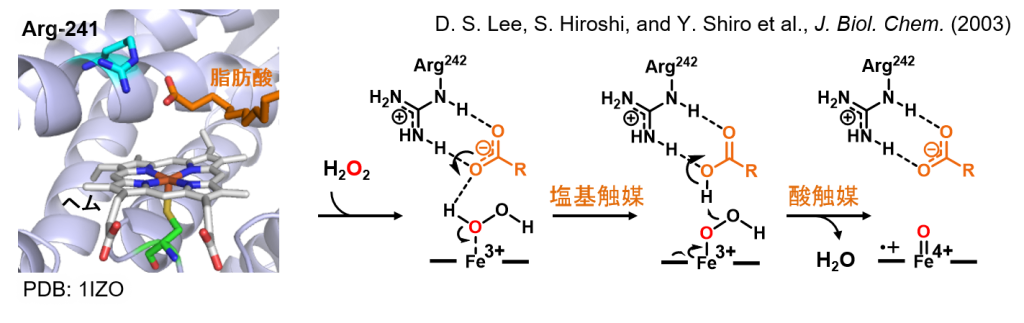
Munroらは、CYP152L1を高純度で精製し、ポリエチレングリコール(PEG)を含む沈殿剤と混ぜる事で、CYP152L1の結晶化に成功しました。得られた結晶は、シンクロトロンと呼ばれる巨大な施設によって生み出されたX-線に照射され、X-線の回折像が検出器に記録されます。数百~数千枚の二次元回折像を解析すると、三次元の電子密度マップが得られ、CYP152L1の構造を確定することができます。
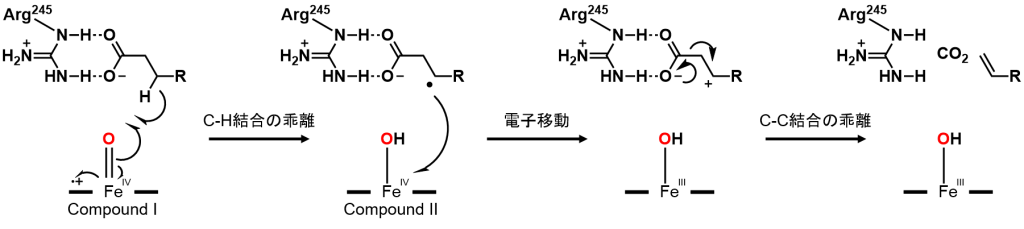
CYP152L1の構造を確認すると、CYP152A1やCYP152B1と同様に、チオレート配位のヘムがCYP152L1の中心に存在し、その真上に脂肪酸が結合していることが判明しました。この構造から、「脂肪酸のβ-炭素がヘムに接近した後、ヘムの酸化活性種によってβ-炭素に結合している水素が引き抜かれ、脱炭酸反応が進行する」という反応機構が提案されました。
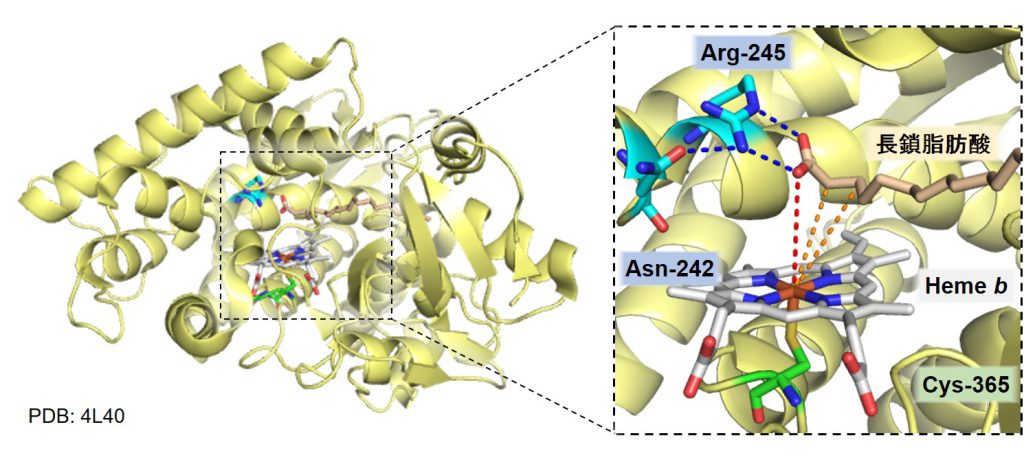
触媒機構の解明
結晶構造によって提案した反応機構を確定する為には、「酸化活性種の観測」や「速度論解析」が必要です。
CYPモノオキシゲナーゼの酸化活性種はCompound Iと呼ばれるオキソフェリルポルフィリンπ-カチオンラジカルであるとされています。CYP152ファミリー酵素は、CYPの中で唯一、天然で過酸化水素を酸化剤として用いる事ができる為、例外的に「CYP152ペルオキシゲナーゼ」とされています。酸化活性種の生成機構が大きく異なるため、酸化活性種がCompound Iであるという事が保証されていませんでした。
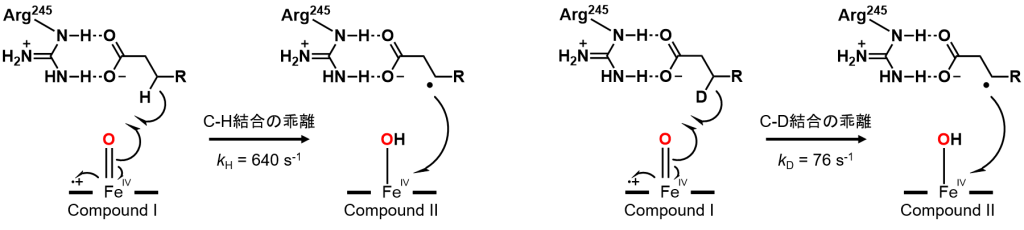
Makrisらは、京都で2014年に開催されたP450の国際学会で、CYP152L1の速度論解析に取り組んでいることを報告していました。2016年、通常の脂肪酸より反応性が低い重水素化脂肪酸とCYP152L1を含む反応溶液中に対して、過酸化水素を加えると、2ミリ秒後にCompound I 特有の370 nmと690 nmの吸収極大波長が観測され、Compound Iが生成していると報告しました。
Compound Iに由来する370 nmの吸収の減少から、Compound Iによる水素(H)の引き抜き速度を見積もると、重水素(D)化脂肪酸では通常の脂肪酸より8倍以上遅い事が判明しました。この現象は速度論的同位体効果(KIE)が現れた場合に生じます。「C-D結合はC-H結合よりも強い」ため、強固なC-D結合の乖離に時間を要し、反応速度が遅くなったと考えられます。この結果から、この脂肪酸脱炭酸反応はC-H結合の乖離によって進行する反応である事が証明されました。
還元酵素との組み合わせ
過酸化水素駆動するCYP152酵素を微生物内で効率的に機能させるためには、培養液に過酸化水素を加える手法や、微生物内で過酸化水素を生産する酵素と共発現させる方法が提案されていますが[22]、活性酸素種の1種である過酸化水素は微生物の生育に悪影響を与えてしまうという問題があります。
Liuらは、P450RhF(CYP116B2)の還元的活性化を担う還元酵素(RhFRED)とCYP152L1の融合タンパク質を大腸菌に発現させることで、過酸化水素非依存的な末端アルケン生成酵素の開発に取り組みました。大腸菌から生成したCYP152L1-RhFRED融合タンパク質は、過酸化水素の添加だけでなく、NADPHの添加によっても、末端オレフィンの生成反応を触媒しました。

基質である脂肪酸を貯め込むように設計した大腸菌 BL21 (DE3) ⊿fatD [pMSD8-ACCase, pMSD15-Thioesterase]株に対して、CYP152L1とCYP152L1-RhFREDを過剰発現することで、炭化水素を生産する大腸菌の作成に取り組みました。残念なことに、CYP152L1-RhFREDを導入した大腸菌よりも、CYP152L1を導入した大腸菌のほうが、末端アルケンの総量が多くなりました(1Lの培養液当たり末端アルケン0.097g)。
大腸菌由来のフェレドキシンとフェレドキシン還元酵素がCYP152L1を還元できた為、RhFREDを融合させていないCYP152が大腸菌中でアルケンを生成できたのでしょう。RhFREDを融合タンパク質として発現する際に、CYP152L1の発現量が低下してしまい、大腸菌中のアルケン生成効率が低下してしまった為、融合タンパク質を発現する手法が有効に機能しなかったとLiuらは考察しています。
CYP152ファミリー酵素が発見された際、CYP152B1の還元的活性化を用いた脂肪酸の酸化反応が進行しなかったことから、これまでCYP152ファミリー酵素は還元的に利用できないと考えられてきました[4]。本研究は、当初の設計どおりには行かなかったもの、CYP152L1が還元的に活性化出来ることを示した最初の論文となります。
中鎖アルケンと短鎖アルケンの生成
鎖長が長い炭化水素は、ガソリン用のエンジンでは発火しづらく、低温では固体になるといった問題から、直接ガソリン車の燃料にとして利用する事が難しいとされています。CYP152L1は脂肪酸からアルケンを生産可能な有用なバイオ酵素として期待されていましたが、Dennigらの報告以前に、CYP152L1は長鎖脂肪酸以外の脂肪酸を扱えないという重大な問題があった為、実用化されないと考えられていました。
CYP152L1が発見されてから10年経過していないのにもかかわらず、CYP152L1を用いた研究が活発になった影響として、還元酵素を用いたシステムでCYP152L1は短い末端アルケンを生産できると示したこの論文が挙げられます。
2011年にCYP152L1を発見したRudeらの報告[10]では、炭化水素を生産する菌とその原因酵素の特定に着目されており、炭化水素生産菌が利用する天然基質に近い長鎖脂肪酸の活性しか調べられておりませんでした。2014年に結晶構造を報告したMunroらの報告[19]やLiuらの報告[21]では、炭素数10の脂肪酸(デカン酸の酸化反応は進行しないと報告されていました。この結果はCYP152B1が炭素数10以下の短い脂肪酸を水酸化できないという松永らの報告[24]と一致しており、炭素数12以上の脂肪酸がCYP152ファミリー酵素の基質寛容性の限界だと考えられていました。
実際、私達の研究室では、CYP152A1やCYP152B1が短い脂肪酸を効率的に酸化できない事を逆手に乗り、炭素数7の脂肪酸(ヘプタン酸)[25]や最も短い脂肪酸(酢酸)[26]存在下で、CYP152酵素を誤作動させ、非天然基質を酸化させるシステムを開発していました。
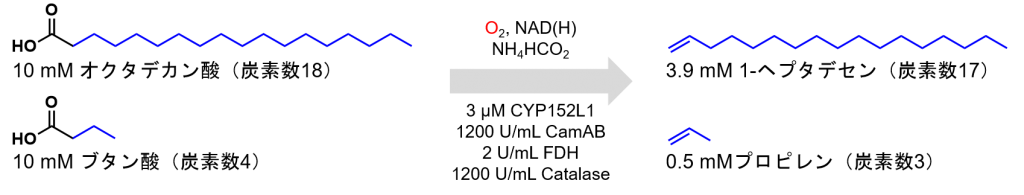
ところが、CYP101A1(P450cam)を還元する酵素(CamAB)を添加したCYP152L1は、炭素数10の脂肪酸(デカン酸)の酸化を効率的に行えませんでしたが、炭素数9~4の脂肪酸の酸化を効率的に行いました。さらにDennigらは、反応条件を調整することで、CYP152L1に炭素数10の脂肪酸を酸化させ、炭素数21~3の末端アルケンを生産させることに成功しました。
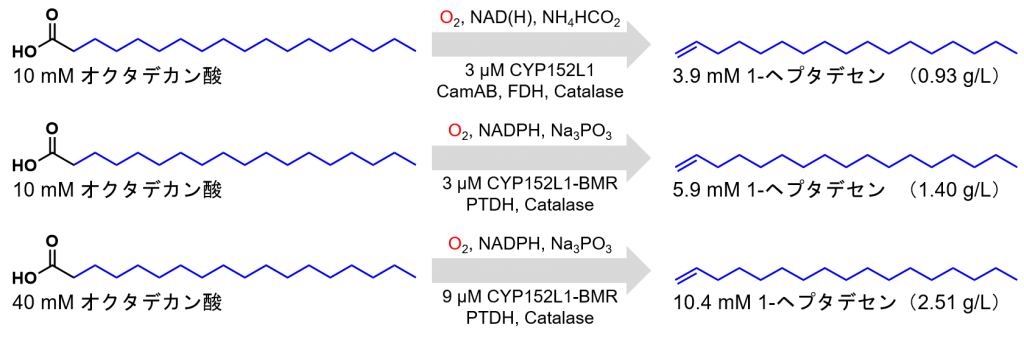
最終的にDennigらは、10 mMの脂肪酸に対して3 μMのCYP152L1を加え、還元剤として200 μMのNAD(H)、還元剤再生剤として100 mMのギ酸アンモニウム、触媒量のCamAB(還元酵素)、ギ酸デヒドロゲナーゼ(NADHの再生酵素)、カタラーゼ(過酸化水素の分解酵素)を加えることで、1Lの反応溶液当たり0.93gのアルケンの生産に成功しています。
収率73%! アルケン生成反応
この論文は、Dennigらの短いアルケンを生成した結果とLiuらの還元酵素との融合タンパク質の設計に影響受けた上で、Dennigらより数倍高い収量に到達したという論文になります。Liuらは、CYP116B2に融合している還元酵素(RhFRED)をCYP152L1に連結しておりました。
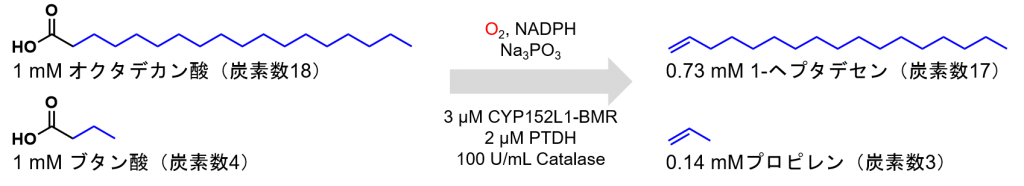
Luらは、最も高い触媒活性を示すシトクロムP450であるCYP102A1(P450BM3)に融合している還元酵素(BMR)を、CYP152L1に連結することで、NADPH依存的なアルケン生産系を立ち上げました。興味深いことに、このCYP152L1-BMR融合タンパク質は24時間室温で静置しても活性低下が見られず、凍結乾燥により粉末状態にして冷凍庫に2ヶ月保存しても活性低下が見られませんでした。
Luらは、1 mMの脂肪酸に対して3 μMのCYP152L1-BMRを加え、還元剤として200 μMのNADP(H)、還元剤再生剤として10 mMの亜リン酸ナトリウム、亜リン酸デヒドロゲナーゼ(NADPHの再生酵素)、カタラーゼ(過酸化水素の分解酵素)を加えることで、炭素数20~6の脂肪酸を酸化して、73%~46%の収率でアルケンの生成に成功しています。
特に最も収率が高かった炭素数18のパルミチン酸の酸化反応では、1Lの反応溶液当たり0.17gのアルケンの生産に成功していましたが、Dennigらの条件と近い基質量を10 mMにした条件では、Dennigらの結果(0.93 g/L)よりも収量が多く、1Lの反応溶液当たり1.40gのアルケンの生産に成功しました。基質の量(40 mM)と酵素の量(9 μM)を最適化した条件では、1Lの反応溶液当たり2.51gのアルケンの生産に成功しました。
脂肪酸の鎖長制御
Dinnigらの論文やLuらの論文よって、CYP152L1は短い脂肪酸を酸化して「バイオガソリンに適切な長さの末端アルケン」を生成出来ると示されましたが、短いアルケンを微生物から低コストに生産する際、微生物中に短い脂肪酸を生産させる必要があります。
Rudeらの論文では、CYP152L1の宿主であるJeotgalicoccus sp. ATCC 8456中の脂肪酸をCYP152L1が脱炭酸することで、炭素数21のイソ脂肪酸から生産された、炭素数20の分岐アルケンが主生成物となり、基質を外部添加しない限り炭素数17以下のアルケンは生産されませんでした。
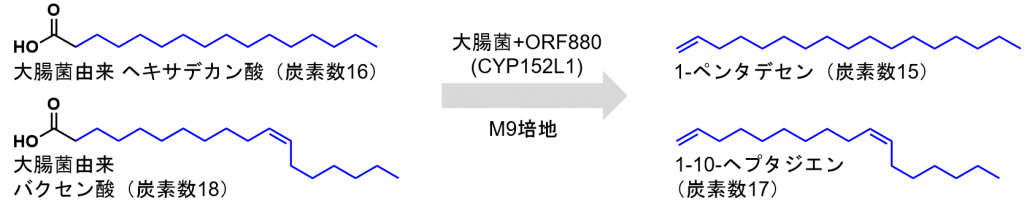
Liuらの論文では大腸菌をCYP152L1に過剰発現させることで、大腸菌中の脂肪酸から末端アルケンを生成しています。Liuらはタンパク質発現用大腸菌BL21 (DE3)に脂肪酸の代謝(β-酸化的分解)を開始させる遺伝子fadDを取り除いた大腸菌株X-100 (DE3) 株を利用し、脂肪酸の生合成に関与するACCaseとチオエステラーゼを過剰発現させることで、菌体中の脂肪酸量を0.23 g/Lに増加させることで、アルケンの生成量を0.097g /Lまで増量させています。
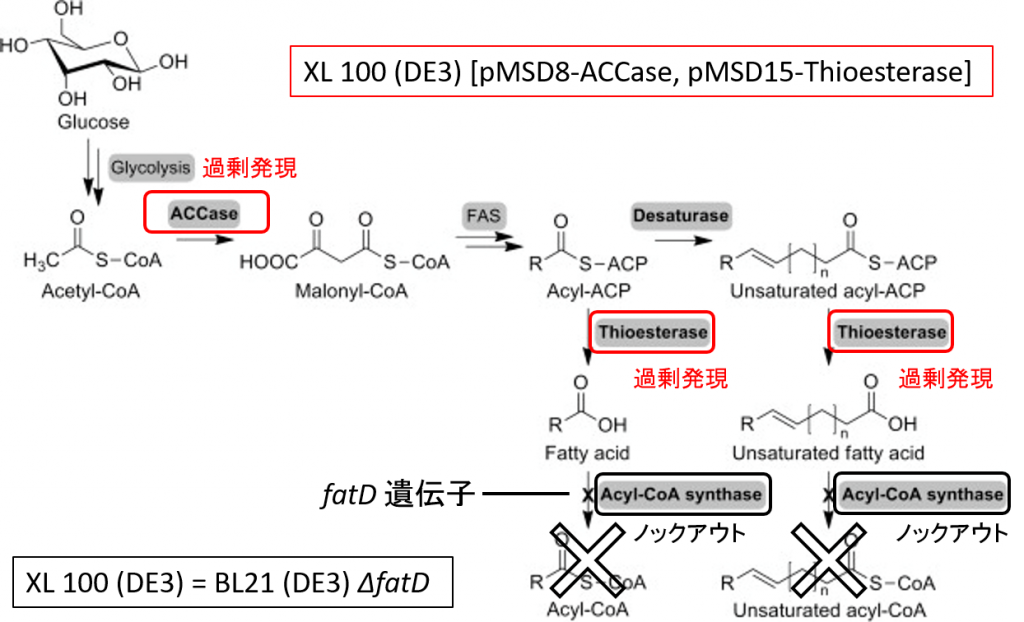
大腸菌の脂肪酸は、炭素数16の飽和脂肪酸(ヘキサデカン酸、通称:パルミチン酸)や炭素数18の不飽和脂肪酸(バクセン酸、cis-11-オクタデセン酸)が大半を占めます。CYP152L1を過剰発現したXL 100 (DE3) [pET28b-CYP152L1]は炭素数17の分岐アルケンや炭素数15のアルケンが主生成物になります。
一方で、CYP152L1を過剰発現させたXL 100 (DE3) [pMSD8-ACCase, pMSD15-Thioesterase, pET28b-CYP152L1]株は、チオエステラーゼが過剰に発現している影響で、ヘキサデカン酸よりも炭素数14の脂肪酸(テトラデカン酸、通称:ミリスチン酸)の存在比が高くなっております。そのため、炭素数13のアルケンと炭素数17の分岐アルケンが主生成物となりました。
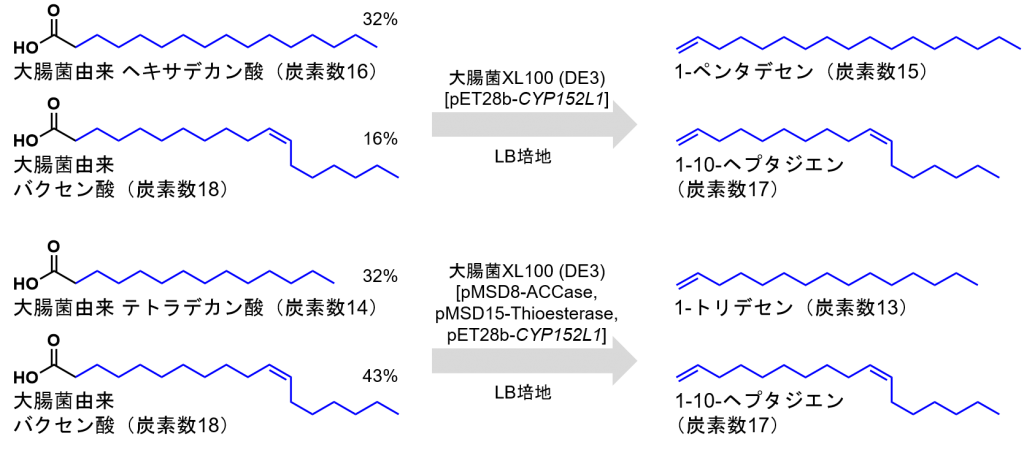
炭素数10以下のの脂肪酸はこのチオエステラーゼから生成されないため、バイオガソリンに適した炭素鎖長のアルケンはほとんど生成していませんでした。そこで、大腸菌に多く存在する炭素数14程度の長鎖脂肪酸から、短い脂肪酸を生産する機構が必要とされていました。
私達は、2013年から好熱性細菌Exiguobacterium sp. AT1b由来のCYP152ファミリー酵素を調べており、このCYP152N1が炭素数14の脂肪酸から炭素数13-9の脂肪酸を生産する事を発見しました[9]。
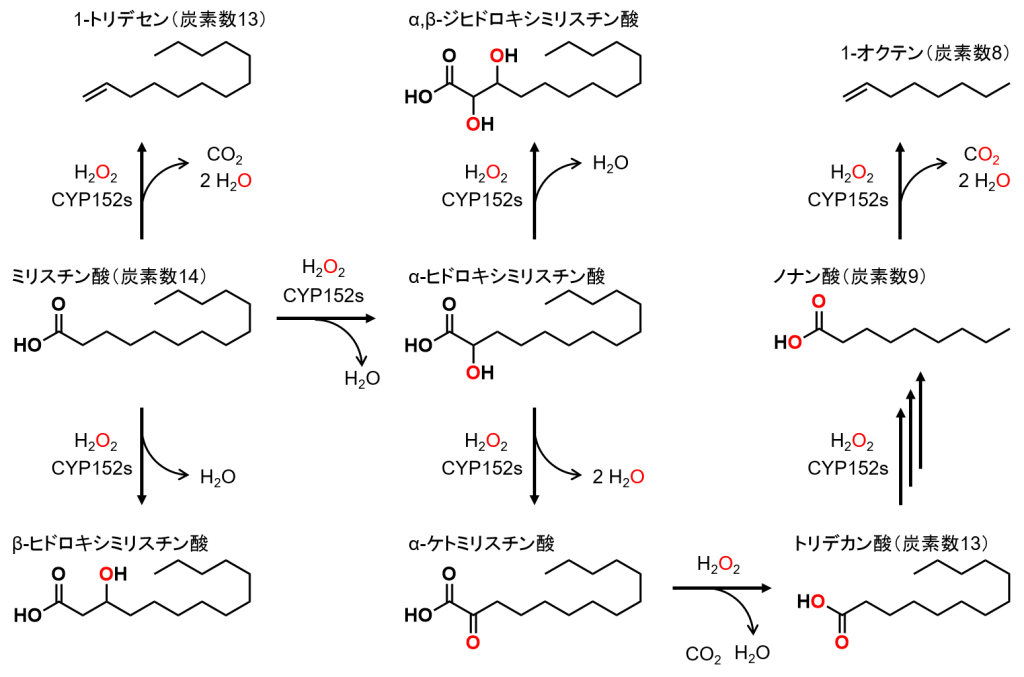
CYP152N1は脂肪酸のα-位選択性が非常に高く、CYP152L1と異なり脂肪酸のβ-位を酸化できないため、β-位の水素引き抜きから進行する末端アルケン生成反応を触媒することができませんでした。通常、CYP152N1は脂肪酸のα-位を(S)-立体選択的に水酸化し、(S)-α-ヒドロキシ脂肪酸を生成しますが、過酸化水素が過剰に存在すると、CYP152N1は生成物をさらに酸化してα-ケト脂肪酸を生成します。このα-ケト脂肪酸が過酸化水素により脱炭酸され、炭素数が1短い脂肪酸が生成していました。
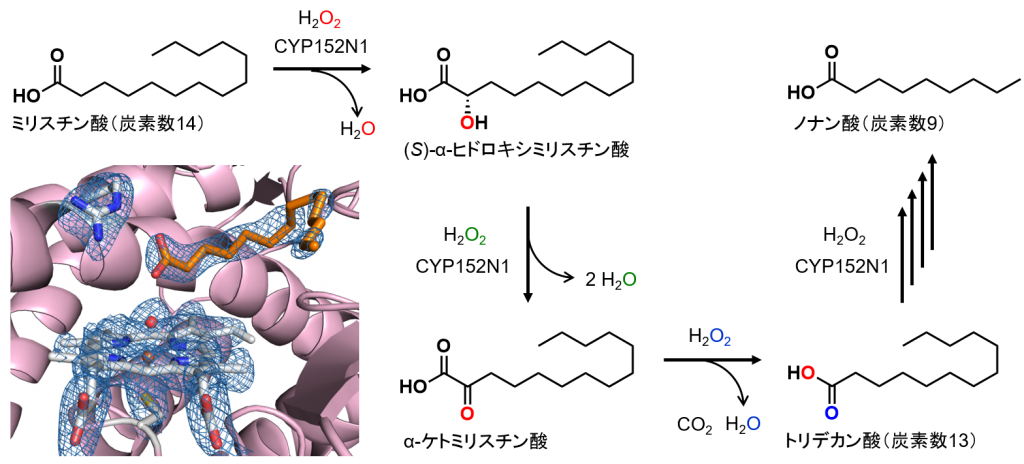
脂肪酸のα-位を(S)-立体選択的に水酸化する反応は、天然でも機能するほど低濃度の過酸化水素(Km < 50 μM)で進行するのですが、(S)-α-ヒドロキシ脂肪酸を過剰酸化してα-ケト脂肪酸を生成する反応は、数mM程度の高濃度な過酸化水素存在下で高い触媒活性を示しました。
高濃度の過酸化水素存在下では、CYP152N1だけでなく、CYP152A1やCYP152B1という一般的なCYP152ファミリー酵素もα-酸化的な脱炭酸反応が進行しました。CYP152A1はわずかに末端アルケン生成反応を触媒するので、この「脂肪酸の炭素鎖を短くするα-酸化的な脱炭酸反応」は「脂肪酸から末端アルケンを生成する反応」と共存出来る可能性を提案できました。
CYP152A1はCYP152N1ほど効率的に鎖長を短くできず、CYP152L1ほど効率的に末端アルケンを生成する事ができないので、短い炭素鎖長の末端アルケンを生産するに至っていません。昨年は3つの新しいCYP152ファミリー酵素[17], [18]が報告され、今年は既に、5つの新しいCYP152ファミリー酵素[28], [29], [30]が報告されています。特に、CYP152P1はγ-、δ-、ε-位の脂肪酸水酸化反応を触媒出来る事が報告され[17]、CYP152K6はα-位とβ-位を同時に水酸化出来ることも報告されました[28]。しかしながら、未だ機能が明らかになっていないCYP152が850種ほどデータベースに登録されております。今後、網羅的にCYP152ファミリー酵素を調べることで効率的に短いアルケンを生成するCYP152が発見されるのではないかと期待しています。
あとがき
微生物に酵素を発現させて目的の物質を生産する「発酵法」は、アルコールやアミノ酸の生産、医薬品中間体の生産に利用されています。このトピックレビューでは、私の研究対象であるCYP152酵素を用いたバイオ燃料の発酵的生産ついてまとめさせていただきました。
私は2013年から学部生としてCYP152N1の研究を遂行していましたが、2014年のMunroらの報告で初めてCYP152L1の存在に気付きました。興味深い研究だと思いましたが、担当教官から「アルケン生産の研究に参入するのではなく、新しい物性や機能を見つけましょう」という提案に賛同し、酵素の誤作動の研究や、脂肪酸の鎖長制御の研究を遂行してきました。最終的には、酵素の誤作動の研究[26]はCYP152L1の活性種寿命測定という研究[31]に貢献し、脂肪酸の鎖長制御の研究[9]はCYP152L1がバイオ燃料生産に利用する原料の鎖長制御を可能とする足がかりになると期待されています。学部生から博士課程までの6年間の研究で、私は周りの面白い研究結果を追随するのではなく、自分の研究の軸をしっかり保つことが大事であると実感しました。
私のバイオ燃料に関わる研究は「脂肪酸の鎖長制御」で終了してしまいましたが、現在もCYP152酵素の研究は続けていますし、就職後もCYPという酵素に関わる仕事が出来るようです。このCYP152酵素を利用したアルケン生産の特許はDennigらの研究を受けてフランスのEvonik社(ボルシア・ドルトムントのスポンサーとして有名)が持っており、今後も研究が続いていくと思われますので、私はフォローし続けてていきたいと思います。
参考文献リスト
- T. Omura, R. Sato, “The carbon monoxide-binding pigment of liver microsomes: I. Evidence for its hemoprotein nature”, J. Biol. Chem. 1964, 239, 2370-2378,
- R. W. Estabrook, D. Y. Cooper, O. Rosenthal, “The light reversible carbon monoxide inhibition of the steroid C21-hydroxylase system of the adrenal cortex.”, Biochem. Z. 1963, 338, 741-755.
- I. Matsunaga, E. Kusunose, I. Yano, K. Ichihara.”Separation and partial characterization of soluble fatty acid α-hydroxylase from sphingomonas paucimobilus“, Biochem. Biophys. Res. Commun. 1994, 201, 1554-1560.
- I. Matsunaga, M. Yamada, E. Kusunose, Y. Nishiuchi, I. Yano, K. Ichihara”Direct involvement of hydrogen peroxide in bacterial α‐hydroxylation of fatty acid”, FEBS Lett. 1996, 386, 252-254.
- E. Yabuuchi, I. Yano, H. Oyaizu, Y. Hashimoto, T. Ezaki, H. Yamamoto, “Proposals of Sphingomonas paucimobilis gen. nov. and comb. nov., Sphingomonas parapaucimobilis sp. nov., Sphingomonas yanoikuyae sp. nov., Sphingomonas adhaesiva sp. nov., Sphingomonas capsulata comb, nov., and two genospecies of the genus Sphingomonas“, Microbiol. Immunol. 1990, 34, 99-119.
- I. Matsunaga, A. Ueda, N. Fujiwara, T. Sumimoto, K. Ichihara, “Characterization of the ybdT gene product of Bacillus subtilis: Novel fatty acid β‐hydroxylating cytochrome P450″, Lipids 1999, 34, 841-846.
- D. S. Lee, A. Yamada, H. Sugimoto, I. Matsunaga, H. Ogura, K. Ichihara, S. Adachi, S. Y. Park, Y. Shiro, “Substrate recognition and molecular mechanism of fatty acid hydroxylation by cytochrome p450 from Bacillus subtilis: Crystallographic, spectroscopic, and mutational studies”, J. Biol. Chem. 2003, 278, 9761-9767.
- T. Fujishiro, O. Shoji, S. Nagano, H. Sugimoto, Y. Shiro, Y. Watanabe, “Crystal structure of H2O2-dependent cytochrome P450spα with its bound fatty acid substrate: insight into the regioselective hydroxylation of fatty acids at the α position”, J. Biol. Chem., 2011, 286, 29941-29950.
- H. Onoda, O. Shoji, K. Suzuki, H. Sugimoto, Y. Shiro, Y. Watanabe, “α-Oxidative decarboxylation of fatty acids catalysed by cytochrome P450 peroxygenases yielding shorter-alkyl-chain fatty acids”, Catal. Sci. Technol. 2018, 8, 434-442.
- M. A. Rude, T. S. Baron, S. Brubaker, M. Alibhai, S. B. D. Cardayre, A. Schirmer, “Terminal olefin (1-alkene) biosynthesis by a novel p450 fatty acid decarboxylase from jeotgalicoccus species” Appl. Environ. Microbiol. 2011, 77, 1718-1727
- A. Schirmer, M. A. Rude, X. Li, “Microbial biosynthesis of alkanes”, Science, 2010, 329, 559-562.
- S. J. Morrison, T. G. Tornabene, W. E. Kloos, “Neutral lipids in the study of relationships of members of the family micrococcaceae.”, J. Bacteriol. 1971, 108, 353-358.
- Y. G. Chen, Y. Q. Zhang, J. X. Shi, H. D. Xiao, S. K. Tang, Z. X. Liu, K. Huang, X. L. Cui, W. J. Li, “Jeotgalicoccus marinus sp. nov., a marine bacterium isolated from a sea urchin”, Int. J. Syst. Evol. Microbiol. 2009, 59, 1625-1629.
- X. Q. Guo, R. Li, L. Q. Zheng, D. Q. Lin, J. Q. Sun, S. P. Li, W. J. Li, J. D. Jiang, “Jeotgalicoccus huakuii sp. nov., a halotolerant bacterium isolated from seaside soil”, Int. J. Syst. Evol. Microbiol. 2010, 60, 1307-1310
- J. H. Yoon, K. C. Lee, N. Weiss, K. H. Kang, Y. H. Park, “Jeotgalicoccus halotolerans gen. nov., sp. nov. and Jeotgalicoccus psychrophilus sp. nov., isolated from the traditional Korean fermented seafood jeotgal”, Int. J. Syst. Evol. Microbiol. 2003, 53, 595-602.
- M. Girhard, S. Schuster, M. Dietrich, P. Dürre, V. B. Urlacher, “Cytochrome P450 monooxygenase from Clostridium acetobutylicum: A new α-fatty acid hydroxylase”, Biochem. Biophys. Res. Commun. 2007, 362, 114-119.
- J. A. Amaya, C. D. Rutland, T. M. Makris, “Mixed regiospecificity compromises alkene synthesis by a cytochrome P450 peroxygenase from Methylobacterium populi“, J. Inorg. Biochem., 2016, 158, 11-16.
- H. Xu, L. Ning, W. Yang, B. Fang, C. Wang, Y. Wang, J. Xu, S. Collin, F. Laeuffer, L. Fourage, S. Li, “In vitro oxidative decarboxylation of free fatty acids to terminal alkenes by two new P450 peroxygenases”, Biotechnol. Biofuels. 2017, 7, 208.
- J. Belcher, K. J. McLean, S. Matthews, L. S. Woodward, K. Fisher, S. E. Rigby, D. R. Nelson, D. , Potts, M. T. Baynham, D. A. Parker, D. Leys, A. W. Munro, “Structure and biochemical properties of the alkene producing cytochrome P450 OleTJE (CYP152L1) from the Jeotgalicoccus sp. 8456 bacterium.”, J. Biol. Chem. 2014, 289, 6535-6550.
- J. L. Grant, M. E. Mitchell, T. M. Makris, “Catalytic strategy for carbon−carbon bond scission by the cytochrome P450 OleT”, Proc. Natl. Acad. Sci. U. S. A. 2016, 113, 10049-10054.
- Y. Liu, C. Wang, J. Yan, W. Zhang, W. Guan, X. Lu and S. Li, “Hydrogen peroxide-independent production of α-alkenes by OleTJE P450 fatty acid decarboxylase”, Biotechnol. Biofuels 2014, 7, 28.
- S. Matthews, K. L. Tee, N. J. Rattray, K. J. McLean, D. Leys, D. A. Parker, R. T. Blankley, A. W. Munro, “Production of alkenes and novel secondary products by P450 OleTJE using novel H2O2‐generating fusion protein systems”, FEBS Lett. 2017, 591, 737-750
- A. Dennig M. Kuhn, S. Tassoti, A. Thiessenhusen, S. Gilch, T. Bülter, T. Haas, M. Hall, K. Faber, “Oxidative decarboxylation of short-chain fatty acids to 1-alkenes.”, Angew. Chem. Int. Ed. 2015, 54, 8819-8822.
- I. Matsunaga T. Sumimoto, A. Ueda, E. Kusunose, K. Ichihara, “Fatty acid‐specific, regiospecific, and stereospecific hydroxylation by cytochrome P450 (CYP152B1) from Sphingomonas paucimobilis: Substrate structure required for α‐hydroxylation”, Lipids 2000, 35, 365-371.
- O. Shoji, T. Fujishiro, H. Nakajima, M. Kim, S. Nagano, Y. Shiro, Y. Watanabe, “Hydrogen peroxide dependent monooxygenations by tricking the substrate recognition of cytochrome P450BSbeta.”, Angew. Chem. Int. Ed. Engl. 2007, 46, 3656-3659.
- H. Onoda, O. Shoji, K. Suzuki, H. Sugimoto, Y. Shiro, Y. Watanabe, “α-Oxidative decarboxylation of fatty acids catalysed by cytochrome P450 peroxygenases yielding shorter-alkyl-chain fatty acids”, Catal. Sci. Technol. 2018, 8, 434-442.
- C. Lu, F. Shen, S. Wang, Y. Wang, J. Liu, W. J. Bai, X. Wang, “An engineered self-sufficient biocatalyst enables scalable production of linear α-olefins from carboxylic acids”, ACS Catalysis 2018, 8, 5794–5798.
- H. M. Girvan, H. Poddar, K. J. McLean, D. R. Nelson, K. A. Hollywood, C. W. Levy, D. Leys, A. W. Munro, “Structural and catalytic properties of the peroxygenase P450 enzyme CYP152K6 from Bacillus methanolicus“, J. Inorg. Biochem. 2018, 188, 18-28
- J. W. Lee, N. P. Niraul, C. T. Trinh, “Harnessing a P450 fatty acid decarboxylase from Macrococcus caseolyticus for microbial biosynthesis of odd chain terminal alkenes”, Metab. Eng. Commun. 2018, 7, 1-8.
- F. Jalu, N. Putra, S. P. Putri, E. Fukusaki, “Metabolomics-based profiling of three terminal alkene-producing Jeotgalicoccus spp. during different growth phase”, J. Biosci. Bioeng. 2018, in press.
- C. H. Hsieh, X. Huang, J. A. Amaya, C. D. Rutland, C. L. Keys, J. T. Groves, R. N. Austin, T. M. Makris, “The enigmatic P450 decarboxylase OleT is capable of, but evolved to frustrate, oxygen rebound chemistry”, Biochemistry 2017, 56, 3347-3357.